В чём сила программы AlphaFold
Белки — это и молекулярные машины, и строительные блоки, и оружие живой клетки. Белки образованы полипептидными цепями, обычно весьма длинными, включающими сотни аминокислотных остатков. Клетка синтезирует белки из двадцати основных аминокислот, последовательность которых в полипептидных цепях кодируется генами. Но ни один белок не существует в виде прямой «палки» из сшитых друг с другом аминокислот. Аминокислотные остатки полипептидной цепи неизбежно взаимодействуют между собой. Физико-химические взаимодействия определяют геометрию связей между атомами, их сближение и отталкивание, их отношения с окружающей средой — водой, ионами, другими молекулами. Полипептидная цепь многократно изгибается, накладывается сама на себя — будучи изначально неупорядоченной, она приобретает определённую, трёхмерную структуру. Происходит то, что называется самоорганизацией, или сворачиванием белка. Именно в свёрнутом виде белок, можно сказать, становится самим собой. Биологические функции белков тесно связаны с тем, как выглядят их трёхмерные (3D) структуры.
Многие тысячи таких пространственных структур уже определены экспериментально — с помощью рентгеноструктурного анализа, ядерного магнитного резонанса и криоэлектронной микроскопии. Эти трёхмерные «портреты» белков хранятся — в виде огромных наборов 3D-координат всех атомов белка — в компьютерном банке белковых данных PDB (Protein Data Bank) и других подобных банках. Однако рентгеноструктурный анализ, ядерный магнитный резонанс и криоэлектронная микроскопия весьма трудоёмки. Поэтому миллиарды белков из самых разных организмов, от вирусов и бактерий до позвоночных, до сих пор остаются с неизвестными пространственными структурами. А их полезно знать — в частности (но не только), для создания новых лекарств.
В то же время для сотен миллионов белков уже известны аминокислотные последовательности их полипептидных цепей, определять которые много легче. Но их простран-ственная структура остаётся тайной.
Поэтому большой интерес вызывают методы предсказания белковых 3D-структур по аминокислотным последовательностям. Как уже было сказано, сворачивание белка происходит благодаря взаимодействию аминокислотных остатков друг с другом, а также и с кофакторами (гемами или другими макромолекулами), и с окружающей средой. Необходимо подчеркнуть, что пространственная структура — результат самоорганизации, то есть структуры белковых цепей определяются самими аминокислотными последовательностями этих цепей (по крайней мере, если речь о глобулярных белках). Напрашивается вывод, что по аминокислотным последовательностям можно предсказать, в какую 3D-форму свернётся полипептидная цепь. Здесь, однако, нужно уточнить, что сворачивание (самоорганизацию) белка можно понимать двояко — как процесс и как результат.
У «средней» белковой цепи есть огромное количество вариантов пространственной структуры, порядка 10100. Перебор всех 10100 альтернатив потребовал бы миллиардов миллиардов лет. Между тем белок сворачивается в одну и ту же уникальную, свойственную ему трёхмерную структуру всего за несколько секунд или минут1. Решая проблему сворачивания в смысле процесса (то, что порой называют «проблемой сворачивания белка первого рода»), мы отвечаем на вопрос, как может белковая цепь чрезвычайно быстро выбрать свою структуру из гигантского набора возможных вариантов.
Если же мы решаем проблему сворачивания в смысле результата (так называемая проблема сворачивания белка второго рода), то отвечаем на вопрос, какую именно структуру приобретёт белковая цепь с конкретной аминокислотной последовательностью.
Долгое время обе проблемы рассматривались как одна: предполагалось, что как только будет ясно «как может», сразу же станет ясно и «какую именно». Однако впоследствии выяснилось, что это две разные проблемы и решаются они по-разному. Поэтому мы можем позволить себе сейчас забыть о «как может» (тем более, что проблема «как может» решена2, и решена она была в нашем Институте белка РАН) и сосредоточиться на втором вопросе — какую пространственную структуру имеет белковая цепь с определённой аминокислотной последовательностью.
Интерес к пространственным структурам белков привёл к начавшимся в 1970-х годах своеобразным «соревнованиям» методов предсказания. С 1994 года такие «соревнования» проводились каждые два года в формате конференций по критической оценке структурных предсказаний, CASP (Critical Assessment of protein Structure Prediction). В этих конференциях участвовали сотни представителей десятков и сотен научных групп из множества стран. Методы предсказаний, использованные в CASP, первоначально базировались в основном на физике белковых цепей и на статистическом анализе уже известных белковых структур и контактов аминокислотных остатков в них.
Качество работы методов предсказания пространственных структур белков повышалось, но медленно, пока в 2018 году не появилась сделанная в Google DeepMind программа AlphaFold, а затем — AlphaFold 23. Эти программы были основаны на «глубоком обучении» многослойных нейронных сетей, и они сразу на голову — а AlphaFold 2 на две головы — превзошли все остальные «предсказывающие» программы.
Яркий успех программы AlphaFold (а затем — и подобных ей) в определении трёхмерных белковых структур очевиден всем, кто работает в этой области науки, но остаётся ряд вопросов. Во-первых, в чём главная причина такого успеха? Во-вторых, чем именно занимается программа AlphaFold? Основаны ли её предсказания 3D-структур на физике полипептидных цепей? Или алгоритм распознаёт 3D-структуры по сходству аминокислотных последовательностей разных цепей друг с другом — тех, для которых 3D-структуры неизвестны, с теми, для которых 3D-структуры уже получены? В-третьих, если структуры получаются из сходства аминокислотных цепей, можно ли с помощью AlphaFold извлекать из них какие-то физические закономерности? И нужны ли в принципе эти закономерности для успешных предсказаний?
Аминокислотные последовательности и структурные сходства
Если трёхмерная структура определяется аминокислотной последовательностью, то сам собой напрашивается вывод, что чем более схожи аминокислотные последовательности двух белков, тем более схожи они будут своими 3D-«портретами». Насколько должны быть похожи аминокислотные цепи для того, чтобы между ними возникла структурная схожесть? Возьмём два полипептида и вытянем их в линию рядом друг с другом, чтобы напротив первой аминокислоты одной цепи стояла первая аминокислота другой цепи. И вот, например, мы видим, что в двадцатом положении в обеих цепях стоит одна и та же аминокислота (скажем, аланин). А в двадцать первом положении аминокислоты разные. С двадцать второй по двадцать четвёртую позицию мы снова видим в обеих цепях одинаковые аминокислоты, например пролин-глицин-серин. Далее мы особого сходства не видим, но зато фрагмент с 30-й по 40-ю аминокислоты первой цепи очень похож на фрагмент с 38-й по 48-ю аминокислоты второй цепи... И так далее.
Такое совмещение полипептидных цепей с поиском сходств и различий в аминокислотной последовательности называется выравниванием. При выравнивании можно сдвигать цепи друг относительно друга и можно в одной цепи пренебречь каким-то участком последовательности, чтобы другой участок лучше совпал по аминокислотам со второй цепью. В таких случаях говорят про выравнивание со сдвигами, вставками и делециями, то есть исключениями небольших фрагментов аминокислотной последовательности. Это кажется подгонкой под ответ, однако на самом деле все такие сдвиги, делеции и вставки ясно видны при сравнении цепей родственных белков — например, глобинов разных животных, так что подобные манипуляции имитируют происходящие в живых организмах мутации и нередко позволяют хоть как-то предсказать неизвестную структуру по уже известной.
Если две последовательности по итогам такого выравнивания совпали в более чем 25% позиций, их трёхмерные структуры будут очень похожи — это эмпирическая закономерность. Если последовательности идентичны менее чем в 20% позиций, трёхмерные структуры будут сильно расходиться. Последовательности с совпадениями между 20 и 25% в смысле 3D-сходства лежат в некой «сумеречной зоне». Конечно, трёхмерную схожесть оценивают не на глаз, а с помощью параметра RMSD (Root Mean Square Deviation, среднеквадратичное отклонение), среднеквадратичной разности координат атомов трёхмерных структур. Мы накладываем две 3D-структуры друг на друга так, чтобы атомы как можно большего числа аминокислотных остатков этих двух полипептидных цепей оказались бы в (почти) одном и том же месте, то есть с почти одинаковыми координатами, или хотя бы в более или менее близком соседстве — и тогда координаты будут отличаться. Чем меньше (в среднем) эти отличия, то есть чем меньше RMSD, тем полнее совпадают пространственные структуры. Опыт показывает, что если последовательности идентичны на 30%, RMSD 3D-структур составляет около 1,4 ангстрема, при идентичности 20% — около 2 ангстрем, а при 15—10% идентичности RMSD становится больше 5—10 ангстрем и приближается к радиусу белка.
Теперь представим, что у нас есть некий белок с известной аминокислотной последовательностью и неизвестной 3D-структурой. Нам всё равно, какая у него аминокислотная последовательность, пусть она будет случайной. И ещё у нас есть база данных белков с известными 3D-структурами и — допустим — столь же случайными последовательностями аминокислот. Одни белки из базы данных по 3D-структурам сильнее похожи своей аминокислотной последовательностью на белок с ещё неизвестной структурой, другие слабее. Вопрос в том, есть ли в базе данных по 3D-структурам белок, у которого последовательность будет на 25% (или хотя бы на 20%) совпадать с последовательностью белка, для которого мы ищем структуру. Говоря иначе, будет ли в наиболее схожих между собой случайных последовательностях хотя бы 20—25% аминокислотных совпадений.
Очевидно, что всё зависит от общего количества последовательностей, которые есть под рукой, то есть от объёма базы данных. Выше мы говорили, что сейчас есть банки данных, в которых собраны последовательности и расшифрованные структуры самых разных белков. Так, в банке PDB в 2020 году было около 150 000 3D-структур. Ещё важно сразу определиться, какого размера последовательности мы сравниваем. Интуитивно понятно, что чем меньше длина полипептидов и чем больше их есть в нашей библиотеке, тем больше вероятность того, что среди них найдутся два похожих. Однако нужно учитывать биологические реалии. Характерный размер белкового домена (компактного и потому способного к самоорганизации куска белковой структуры) — около 100 аминокислот; этой длиной и стоит оперировать в вероятностных расчётах. И поскольку речь идёт о сходстве в последовательностях аминокислот, нужно уточнить, что такое случайная последовательность. Полипептидные цепи состоят из двадцати аминокислот; следовательно, если речь о случайной цепи, то вероятность любой из этих двадцати аминокислот в любой позиции цепи составляет примерно 1/20. (Известно, что аминокислотные последовательности водорастворимых — наиболее часто встречающихся — глобулярных белков весьма похожи на «случайные» — в них нет ни статистически заметной периодичности, ни заметных сгущений каких-то аминокислот.)
Можно математически показать, что если у нас в банке данных есть 150 000 «случайных» последовательностей из примерно 100 аминокислот каждая, то для той из них, что наиболее похожа на последовательность нашего белка с неизвестной 3D-структурой, доля совпадающих аминокислот обычно составляет 19%.
Если мы имеем в виду поиск структуры по сходству, то 19% — это вблизи «сумеречной» зоны, в смысле надёжности предсказания. На самом же деле проценты аминокислотного сходства получаются несколько больше. Мы уже говорили, что при выравнивании полипептидных цепей можно сдвигать их друг относительно друга, делать вставки и делеции. То есть две цепи можно сравнить, как они есть, без всяких сдвигов (и получить 19% сходства), а можно сравнить со сдвигом, да ещё с делециями в каждой из цепей (и тогда получить 25% сходства). Биологически обе цепи остаются теми же белками, но с точки зрения биоинформатики они разные — иными словами, варианты выравнивания как бы увеличивают объём банка данных. А чем больше банк, тем больше доля совпадающих аминокислот в нашем белке с неизвестной 3D-структурой и наиболее похожей на него последовательностью из банка.
И тогда, если мы ищем наилучшее сходство с нашим белком (3D-структура которого неизвестна) в белковом банке 3D-структур PDB, это сходство вырастет с 19 до 25% — для домена длиной в сто аминокислот. А если взять «полудоменную» последовательность, доля совпадающих аминокислот — с учётом возможностей сдвигов, делеций и вставок — вырастет до 36%, и тем меньше получатся цифры RMSD, указывающие на расхождение пространственных структур, а сами структуры будут более похожи друг на друга.
Но что, если в банке белковых структур не нашлось цепи, похожей на ту, структуру которой мы пытаемся предсказать? Или, иными словами, доля идентичных аминокислот при выравниваниях никак не превысит 19—25%? В таком случае нашу цепь с неизвестной структурой можно сравнивать по частям. Пусть для белка (или домена) в сто аминокислот не нашлось ничего подходящего — эти сто аминокислот можно разделить на два «полудомена», и для каждого из них в банке найдётся достаточно похожий отрезок с известной 3D-структурой. Эти отрезки будут из разных белков, но их структуры алгоритм AlphaFold вполне может состыковать, и тогда мы получим единый 3D-«портрет» для того белка, структуру которого пытаемся предсказать.
Понимает ли искусственный интеллект физику?
Повторим ещё раз: трёхмерная структура определяется аминокислотной последовательностью, а объёмы современных библиотек со структурами достаточно велики, чтобы предсказать неизвестную структуру сугубо биоинформатическими методами. То есть достаточно знать только последовательность аминокислот, не утруждая себя погружением во взаимодействия атомов, в параметры их притяжения, отталкивания и т. д. Программа AlphaFold как раз и занимается биоинформатическим поиском — после соответствующего «глубокого обучения». Обучение формирует набор параметров, которые наиболее нужны при анализе аминокислотных последовательностей и структур. AlphaFold 2 содержит примерно 21 миллион «выученных» параметров нейронной сети — это в 1000 раз больше, чем число параметров, необходимых для описания физики белковых цепей, которая оперирует парными, тройными и четверными взаимодействиями атомов. Можно сказать, что доля чисто биоинформатических усилий, когда структура предсказывается только по сходству аминокислотных последовательностей, в тысячу раз превосходит долю усилий, которые опираются на физику белковых цепей.
Стоит уточнить, что 21 миллион параметров — это те, которые получены как искусно сделанный экстракт, на обучающем материале, то есть на всех имеющихся базах данных. Они образуют собственную, или «внутреннюю», память программы AlphaFold. Но она может использовать и «внешнюю память», то есть полные белковые базы данных со всей имеющейся там информацией. А в PDB, с его 150 000 трёхмерных структур, — порядка миллиарда атомных координат сотни миллионов аминокислотных остатков. А ведь есть ещё банк UniProtKB — в нём хранится более 200 миллионов последовательностей, содержащих почти 100 миллиардов аминокислотных остатков. Всеми этими миллиардами AlphaFold может пользоваться, анализируя всевозможные взаимные контакты аминокислотных остатков. AlphaFold может работать и без этой «внешней памяти», но с нею предсказания получаются лучше и надёжнее4.
Может ли AlphaFold узнать что-то из физики, просто анализируя последовательности и соответствующие им структуры? И нужна ли вообще искусственному интеллекту этой программы физика? Да, из физики AlphaFold кое-что знает. Белковые структуры складываются из разных элементов, которые бывают более или менее стабильными именно с физической точки зрения. Об относительной их стабильности можно судить по статистике, с которой они встречаются в различных белках, — а такую статистику можно извлечь из PDB. Но AlphaFold ничего (пока?) не знает о процессе сворачивания белка. Выше мы говорили, что сворачивание можно понимать в смысле процесса и в смысле результата. И, например, время сворачивания полипептидной цепи (которое относится именно к процессу) AlphaFold предсказать (пока?) не может.
И даже если говорить о предсказании готовых структур (то есть результата сворачивания), можно найти прецеденты, когда AlphaFold явно демонстрирует незнание физики. В частности, алгоритм AlphaFold вполне способен выдать структуру, полностью противоречащую физике этой структуры. При этом сама структура будет вполне правильной, просто для того, чтобы она существовала, её должны поддерживать взаимодействия других полипептидных цепей. Из чисто биоинформатических данных AlphaFold строит именно ту структуру, которая и должна быть. Но искусственному интеллекту не приходит на ум, что сама по себе такая структура существовать не может, — просто потому, что AlphaFold не анализирует физические взаимодействия.
Наконец, есть примеры специально сконструированных белков, в которых мутация одного-единственного определённого аминокислотного остатка кардинально меняет структуру. В этом случае, если попросить AlphaFold предсказать мутантную структуру, то наверняка возникнут проблемы — опять же потому, что алгоритм будет полагаться не на универсальную по своей природе физику, а на данные, хранящиеся в имеющихся на сей день огромных, но всё же ограниченных базах. Тут уместно пояснить, почему «чисто физические» расчёты не помогают справиться с задачей предсказания 3D-структур белков. Сейчас физика может оценивать энергии белковых структур с неизбежными, пусть очень небольшими погрешностями. Эти погрешности достаточно малы, чтобы практически точно, с уверенностью 99,9%, ответить на вопрос: «Какая из двух структур стабильнее, то есть имеет более низкую энергию?» Но эти же погрешности оказываются слишком большими, чтобы ответить на вопрос: «Какая из миллиардов структур самая стабильная?» И вот тут-то срабатывают все гигабайты биоинформатических данных.
В целом можно сказать, что AlphaFold опирается преимущественно на биоинформатику, а своему успеху программа обязана огромным, собранным за полвека трудом сотен тысяч учёных библиотекам белковых данных (и, разумеется, мастерству программистов). Если бы нынешний алгоритм AlphaFold появился в 1974 году, когда были известны примерно тысяча аминокислотных последовательностей и всего около десятка белковых структур, он просто не смог бы работать. Опираясь только на биоинформатические данные, AlphaFold лишь в небольшой степени использует физику белковой цепи и в целом вполне обходится без неё.
***
В заключение можно задаться философскими вопросами. Означает ли хорошее предсказание, что предсказатель понимает, что происходит? Позволяет ли верное понимание того, что происходит, хорошо предсказать результат того, что происходит? Ответ на оба эти вопроса — нет! И хорошую иллюстрацию здесь даёт история астрономии.
Жрецы Древнего Египта и Вавилона неплохо предсказывали затмения Солнца и Луны — потому что у них были огромные архивы за 2 тысячи лет наблюдений. И они знали: если на восходе Солнца молодая Луна была там-то, через месяц Солнце затмится — потому что именно так уже было много раз. А если на заходе Солнца старая Луна была там-то, через полмесяца затмится Луна — опять же, потому что именно так уже было много раз!
Предсказание было точным — при полностью неверном понимании ситуации: жрецы Древнего Египта и Вавилона считали Землю плоской, а о вращении Земли вокруг Солнца и Луны вокруг Земли речи вообще не шло...
Позже Птолемей создал (тоже — располагая огромными архивами) теорию эпициклов, предсказывающую не только затмения Солнца и Луны, но и движение всех известных тогда планет на земном небе. При этом для него и Солнце, и Луна, и все планеты двигались вокруг Земли... Опять — отличное предсказание при неверном понимании ситуации.
Затем Коперник понял, что всё вращается вокруг Солнца, — но постулировал, что все орбиты круговые. И при верном общем понимании процесса Коперник предсказывал движения планет хуже, чем Птолемей (что вредило принятию гелиоцентризма не меньше, чем церковь). А всё из-за численной ошибки: Коперник рассматривал орбиты как круги (имеющие нулевой эксцентриситет), а не как эллипсы — пусть с небольшим, но не нулевым эксцентриситетом. И для некоторых планет Копернику всё же приходилось обращаться к помощи эпициклов, фактиче-ски моделирующих эллиптичность орбит. До эллипсов додумался только Кеплер, а позже Ньютон нашёл причину их существования на основе своей теории гравитации.
И только тогда верное понимание объединилось с верными предсказаниями движений — и не только планет, но и комет, и ракет.
Итак, хорошее предсказание — далеко не гарантия верного понимания, а верное понимание того, что происходит, не обязательно даёт хорошее предсказание при неточных параметрах расчёта. И это в полной мере относится к искусственному интеллекту в лице AlphaFold, программы-предсказателя структур белковых цепей.
Авторы благодарны Н. В. Довидченко, С. О. Гарбузинскому, М. А. Пак, Г. Вринду (G. Vriend) и особенно Дж. Джамперу (J. Jumper) за обсуждение затронутых в статье тем.
Работа поддержана Российским научным фондом (проект № 21-14-00268).
Аминокислоты, пептиды, белки
Полипептидная цепь — это последовательность аминокислот (или, точнее, аминокислотных остатков), соединённых в том порядке, в каком это записано в гене конкретного белка (глобина, инсулина, пищеварительного фермента пепсина и т. д.). Последовательность аминокислот называется первичной структурой белка. Взаимодействуя между собой, аминокислотные остатки полипептидной цепи образуют элементы вторичной структуры, которые потом объединяются в третичную структуру. Не вдаваясь в подробности, можно сказать, что элементы вторичной структуры охватывают более или менее протяжённые участки последовательности внутри полипептидной цепи, тогда как третичная структура охватывает всю полипептидную цепь. Однако в крупных белках нередко бывает так, что отдельные их части приобретают третичную структуру независимо друг от друга. В таком случае мы имеем дело с доменами белка, где каждый домен — независимый, компактный и стабильный элемент «большой» третичной структуры. Домены можно сравнить с крупными комками, сделанными из одной и той же нити. В многодоменных белках функции всей молекулы в целом зависят от взаимодействия доменов между собой. (Некоторые белки, например, гемоглобин, образуются при соединении нескольких отдельных белков, или белковых субъединиц, — в таких случаях говорят уже о четвертичной структуре.)
Третичная структура внешне может выглядеть как нечто округлое, картофелеобразное, с мягкими неровностями и, бывает, торчащими петлями полипептидной цепи. Белки такого пространственного вида называют глобулярными. К ним относится большинство белков, как свободноплавающих в растворе (водорастворимых), так и тех, которые включены в клеточные мембраны (рецепторы, ионные каналы и др.). Важную — в основном структурную — роль играют также фибриллярные (нитеобразные) белки, образующие огромные нерастворимые в воде агрегаты. Наконец, есть нативно-неупорядоченные белки — стабильная пространственная структура образуется у них лишь при взаимодействии с другими макромолекулами.
Нейросеть для всех белков
Машинные нейросети копируют некоторые принципы работы настоящих нейросетей, только нейронами в них служат элементы алгоритма. Между «нейронами» нейросетей есть связи, которые, условно говоря, могут быть сильнее или слабее. Свойства связей описываются весовыми коэффициентами. При глубоком обучении «нейроны» поэтапно ищут взаимные корреляции между разнородной информацией, вычленяя параметры, которые позволяют систематизировать данные. Эти параметры отражаются в весовых коэффициентах связей.
В случае нейросети AlphaFold, предсказывающей структуру белка, весовые коэффициенты «помнят», как часто всевозможные данные об аминокислотной последовательности связаны со структурными элементами. Когда нейросеть получает для предсказания некую последовательность аминокислот, она находит наиболее вероятные расстояния между ними, опираясь на огромное количество прецедентов, сведения о которых преобразовались в весовые коэффициенты связей между «нейронами». Эти предсказанные расстояния (или контакты) описывают структуру белка.
На одно предсказание у AlphaFold уходит 10—20 секунд. В соревновании CASP14 в 2020 году вместе с версией AlphaFold 2 участвовали ещё 146 методов предсказания 3D-структур, и качество предсказаний AlphaFold 2 было как минимум в 2,5 раза выше, чем у прочих алгоритмов — это значит, что предсказанные AlphaFold 2 структуры лучше совпадали с экспериментальными данными. В июле прошлого года разработчики AlphaFold сообщили, что их детище предсказало более 200 млн белковых 3D-структур — почти все известные на сегодняшний день белки, от бактериальных до человеческих.
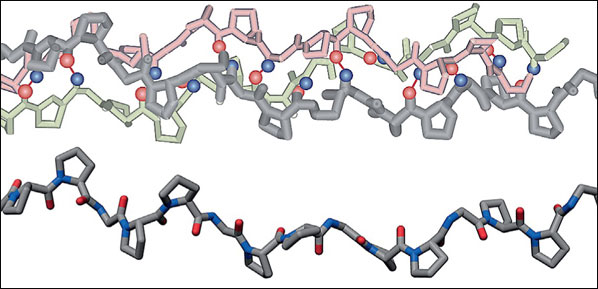
Тем не менее не стоит забывать, что структура, верно предсказанная по биоинформатическим данным, не обязательно существует с точки зрения физики. На рисунке показана пространственная структура белка коллагена (вверху) и предсказанная программой AlphaFold пространственная структура коллагеноподобной полипептидной цепи. Обогащённая аминокислотами глицином (Gly) и пролином (Pro), полипептидная цепь коллагена сворачивается в спираль, которая с двумя такими же спиралями образует тройную супер-спираль. В скелетной модели фрагмента коллагеновой суперспирали (на рисунке — вверху) каждая из трёх спиралей выделена своим цветом. Эту структуру скрепляют водородные связи между NH-группами (синие шарики) и кислородами (красные шарики) и тесные взаимодействия между всеми тремя цепями.
Коллагеноподобный полипептид состоит из повторяющихся троек аминокислот Gly-Pro-Pro. По аминокислотной последовательности цепь должна иметь коллагеноподобную структуру, и AlphaFold совершенно правильно показывает такую структуру для отдельной цепи (предсказанная структура внизу соответствует серой цепи коллагена из настоящей коллагеновой суперспирали вверху). Однако отдельная цепь в таком виде существовать не может, её должны поддерживать взаимодействия с двумя другими такими же цепями. Если бы алгоритм AlphaFold понимал физику взаимодействий аминокислотных остатков, он бы выдавал сообщение, что структура невозможна, либо по собственному почину дорисовал бы дополнительные цепи.
Подробности для любознательных
Полипептидные вероятности
Возьмём случайную последовательность аминокислотных остатков Sn и сравним её с другими случайными последовательностями той же длины, общим числом N. Пусть все эти цепи, включая Sn, состоят из n аминокислотных остатков (для белкового домена характерно n ≈ 100). Каждый тип аминокислоты в каждой позиции выпадает с вероятностью p ≈ 1/20.
Вероятность того, что случайная аминокислотная последовательность Sn совпадает с другой случайной последовательностью той же длины n в m позициях, согласно распределению Пуассона, равна
Pm,p,n = [ (pn)m/m! ] e-pn,
когда каждый тип аминокислотного остатка выпадает с вероятностью p.
Если же случайную последовательность Sn сравнивать не с одной, а с N случайными последовательностями той же длины n, то Pm,p,n • N — ожидаемое число членов из набора случайных последовательностей, совпадающих с Sn в m позициях. Таким образом, уравнение
Pm,p,n • N = 1
определяет максимальное ожидаемое число M совпадений последовательности Sn с наиболее сходной с ней последовательностью из набора N случайных последовательностей.
Если p « 1, а последовательности достаточно длинные (1 « pn), то, интересуясь только парами последовательностей с высокой идентичностью (1 « pn < m), можно использовать приближение Стирлинга m! ≈ (m/e)m, где e ≈ 2,72 и получать
Pm,p,n ≈ (ep/(m/n))m e-pn.
Из этого уравнения следует максимальное ожидаемое значение m/n, обозначаемое как M/n:
((M/n)/pe)•ln((M/n)/pe) = (1/npe)•ln(N) - 1/e.
При p ≈ 1/20, n ≈ 100 и N ≈ 150 000 (столько было 3D-структур в банке PDB в 2020 году, когда появилась программа AlphaFold 2) M/n ≈ 19%, а при N ≈ 200 000 000 (столько было аминокислотных последовательностей в UniProtKB, банке первичных структур белков, в 2020 году) M/n ≈ 24%. И это — без сдвигов, делеций и вставок.
Комментарии к статье
1 Почти мгновенный выбор единственной нужной структуры из колоссального количества возможных называется парадоксом Левинталя, по имени молекулярного биолога Сайруса Левинталя, который сформулировал его в 1968 году. На решение этого парадокса потребовалось почти 30 лет.
2 Финкельштейн А. В., Бадретдинов А. Я. Физические причины быстрой самоорганизации стабильной пространственной структуры белков: Решение парадокса Левинталя. Мол. биол., 31, 469—477 (1997).
3 Senior et al., Proteins 87, 1141—1148 (2019); Jumper et al., Nature 596, 583—589 (2021).
4 Ivankov D., Pak M., Finkelstein A. AlphaFold: predicts or recognizes the protein structure? PROGRAM of the XXVIII Symposium on Bioinformatics and Computer-Aided Drug Discovery, May 24-26, 2022, Moscow (http://www.way2drug.com/dr/bcadd2022_program.php).




